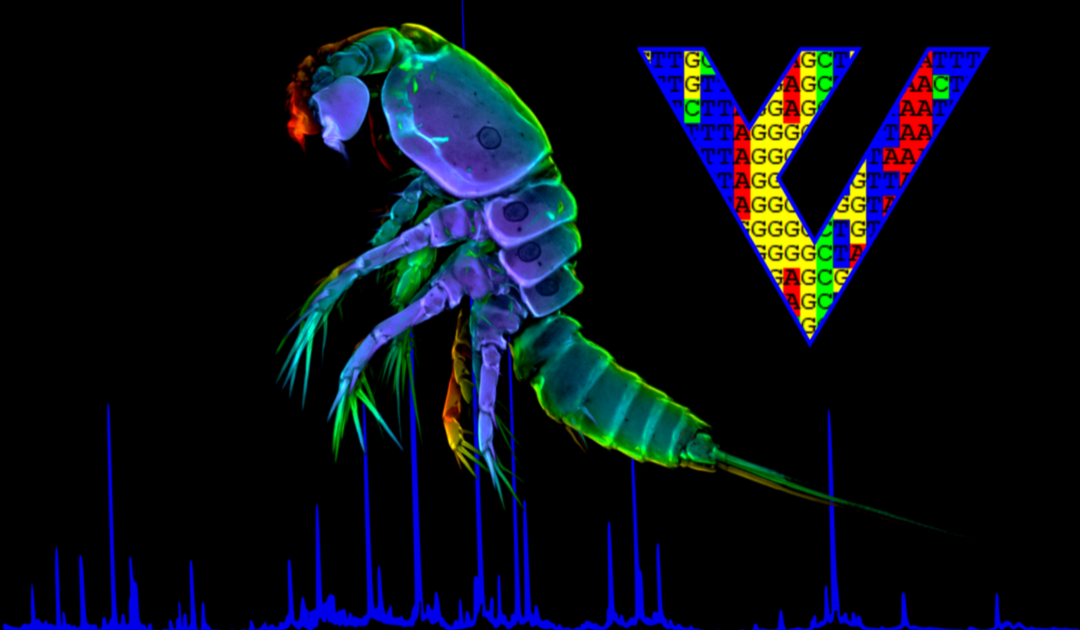
Proteomic Laboratory
Artidentifikation ist eine wesentliche Grundlage ökologischer und wissenschaftlicher Studien. Gleichzeitig ist sie zeitaufwändig und es ist ein hohes Maß taxonomischen Wissens notwendig. Mit Hilfe des Proteom Fingerprintings lassen sich Arten auch ohne jahrelang gesammelte Expertise schnell, günstig und verlässlich identifizieren. In unserem Labor arbeiten wir für die Ermittlung des Proteinfingerabdrucks mit der MALDI-TOF Massenspektrometrie.
MALDI-TOF MS steht für Matrix Assissted Laser Desorption/Ionization Time Of Flight Mass Spectrometry. Bei dieser Methode werden die zu analysierenden Stoffe in eine Matrix eingebettet und mit einem Laser ionisiert. Dadurch werden sie aus der Matrix gelöst, durch ein elektromagnetisches Feld je nach Größe unterschiedlich schnell beschleunigt und am Ende von einem Detektor gemessen. Dies ermöglicht die Messung von Makromolekülen wie Peptide und Proteine, ohne dass diese durch die Strahlung zerstört werden. Die Gesamtheit der gemessenen Peptide und Proteine ergibt dann den Protein-Fingerabdruck. Im Bereich der Mikrobiologie ist das Messen des Protein-Fingerabdrucks bereits seit Langem eine Standardmethode zur Identifikation verschiedener Arten von Bakterien, Pilzen und Viren. In den letzten Jahren hat diese Methode auch Anwendung zur Identifikation von Tieren gefunden.
In unserer Sektion arbeiten wir daran diese Methode für die Anwendung in der Biodiversitätsforschung vorzubereiten. Dafür testen wir diese Methode zunächst an verschiedenen Tiergruppen. Darüber hinaus erarbeiten wir Arbeitsabläufe, die das schnelle Analysieren von MALDI-TOF MS Daten ermöglichen. Insbesondere in Gemeinschaftsanalysen ist es die morphologische Identifikation vieler Individuen, die einen großen Teil der Zeit in Anspruch nimmt. Mit der MALDI-TOF Massenspektrometrie soll diese Zeit deutlich verkürzt werden, da im Vergleich zu anderen Methoden wie dem COI Barcoding nur wenige Präparationsschritte von Nöten sind. Neben dem zeitlichen Vorteil, entstehen bei der MALDI-TOF Massenspektrometrie auch deutlich geringere Kosten, wodurch sie gerade bei großen Indivduenzahlen attraktiv ist. Dadurch bietet sich diese Technik besonders zur Anwendung im Bereich der Meiofaunaforschung sowie der Analyse von Plankton-Lebensgemeinschaften und Zeitreihen an. Darüber hinaus konnten wir auch schon kryptische Arten, also Arten die morphologisch nicht zu unterscheiden sind, identifizieren. Damit ist die Methode auch im Bereich Makro- und Megafauna durchaus interessant und kann zur Unterstützung bei morphologisch schwer zu identifizierenden Arten herangezogen werden, ohne dass es einer langwierigen Vorbereitung der Proben benötigt.