Proteomic Laboratory
Forschung
Voraussetzungen zur Probenbearbeitung
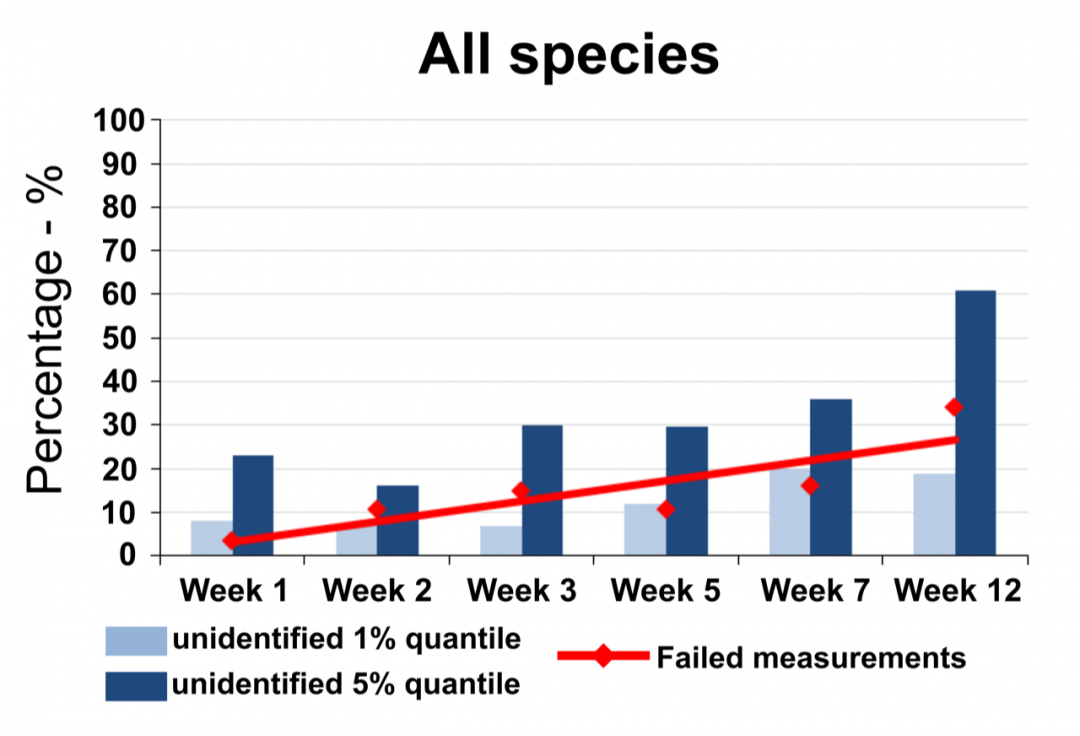
Nicht alle Proben eignen sich zur Bearbeitung mit der MALDI-TOF Massenspektrometrie. Bestimmte Fixative sowie Lagerungsmethoden können ein erfolgreiches Messen des Protein-Fingerabdrucks verhindern. Eine wichtige Aufgabe unseres Labors ist es daher zu ermitteln wie Proben behandelt werden müssen, damit eine erfolgreiche Identifikation auf Basis von Massenspektrometriedaten möglich ist. Dafür wurden bereits umfangreiche Untersuchungen an Meiofauna aus Sedimentproben durchgeführt.
Die Untersuchung von Proben, die mit verschiedenen Ethanolkonzentrationen fixiert sowie bei unterschiedlichen Bedingungen gelagert wurden, zeigte dabei einen starken Einfluss der Lagertemperatur auf die resultierende Qualität der Massenspektren. Ethanolfixierte Proben die bei niedrigen Temperaturen (- 25°C) gelagert wurden zeigten im Vergleich zu frischen Proben nur eine geringe Veränderung der Signalqualität. Eine Lagerung bei Raumtemperatur führte hingegen schon nach kurzer Zeit zu einer deutlichen Abnahme der Signalqualität. Die führte bereits nach wenigen Wochen dazu, dass sich Proben nicht mehr messen ließen.
Bisher werden alle in unserem Labor bearbeiteten Proben mit hochprozentigem Ethaonol fixiert. Weitere Untersuchungen werden zeigen, ob sich auch andere gängige Fixative für das Fixieren von Proben eignen, die für die MALDI-TOF Massenspektrometrie vorgesehen sind.
Pilotstudien an verschiedenen Arten
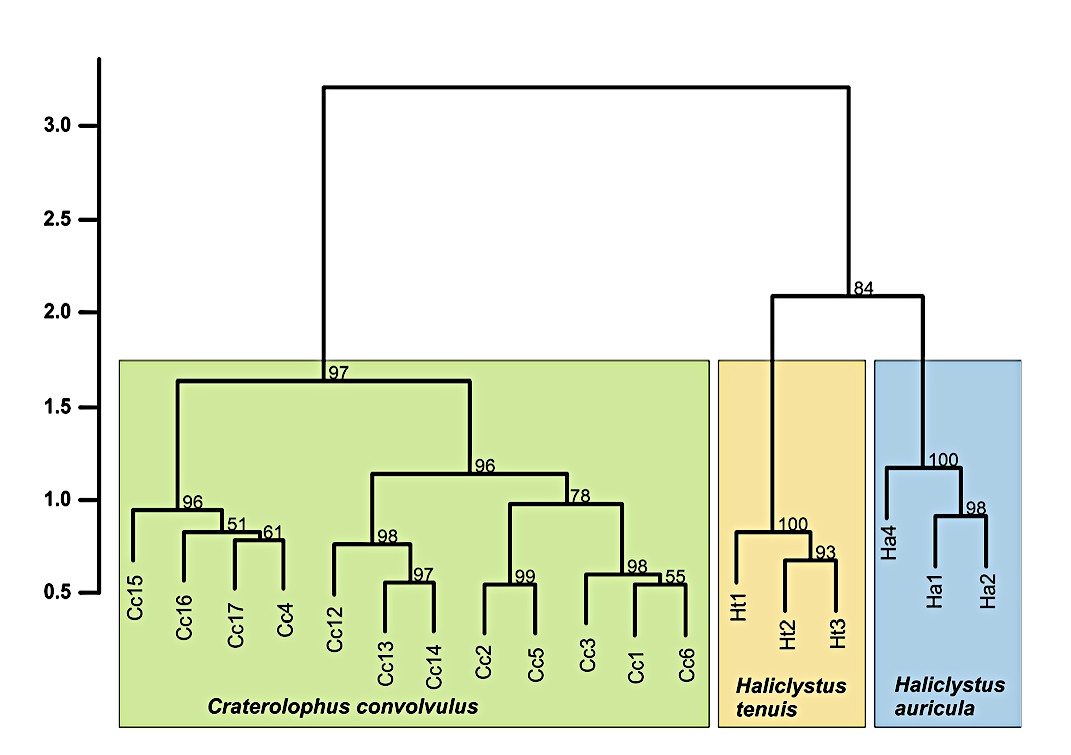
Da der Einsatz der MALDI-TOF Massenspektrometrie für die Identifikation von Tieren noch nicht weit verbreitet ist, muss für die meisten Tiergruppen noch ermittelt werden, ob sich diese Methode erfolgreich anwenden lässt. Dazu werden die Tiere zunächst morphologisch und molekulargenetisch zu identifiziert und es wird geprüft inwiefern sich ontogenetische oder andersartige Unterschiede auf die Identifikation mittels Massenspektrometrie auswirken. In unserem Labor werden hauptsächlich marine Organismen untersucht.
Wir konnten bereits zeigen, dass sich calanoide Copepoden der Nordsee leicht anhand eines Proteinfingerabdrucks unterscheiden lassen und sogar die Differenzierung von Larvenstadien möglich ist. Auch für die deutlich kleineren harpacticoiden Copepoden der Nordsee war die Bearbeitung mittels MALDI-TOF MS bereits erfolgreich. Darüber hinaus konnten auch größere Tierarten wie Cnidaria (Nesseltiere), deren morphologische Identifikation sehr schwierig ist, schnell und verlässlich identifiziert werden.
Durch vorangegangene Projekte am DZMB (MOLTAX Arbeitsgruppe) steht uns eine große Gewebedatenbank morphologisch und molekulargenetisch bestimmter Organismen der Nordsee zur Verfügung, deren Bearbeitung mit dem MALDI-TOF noch aussteht.
Methoden zur schnellen Biodiversitätserfassung
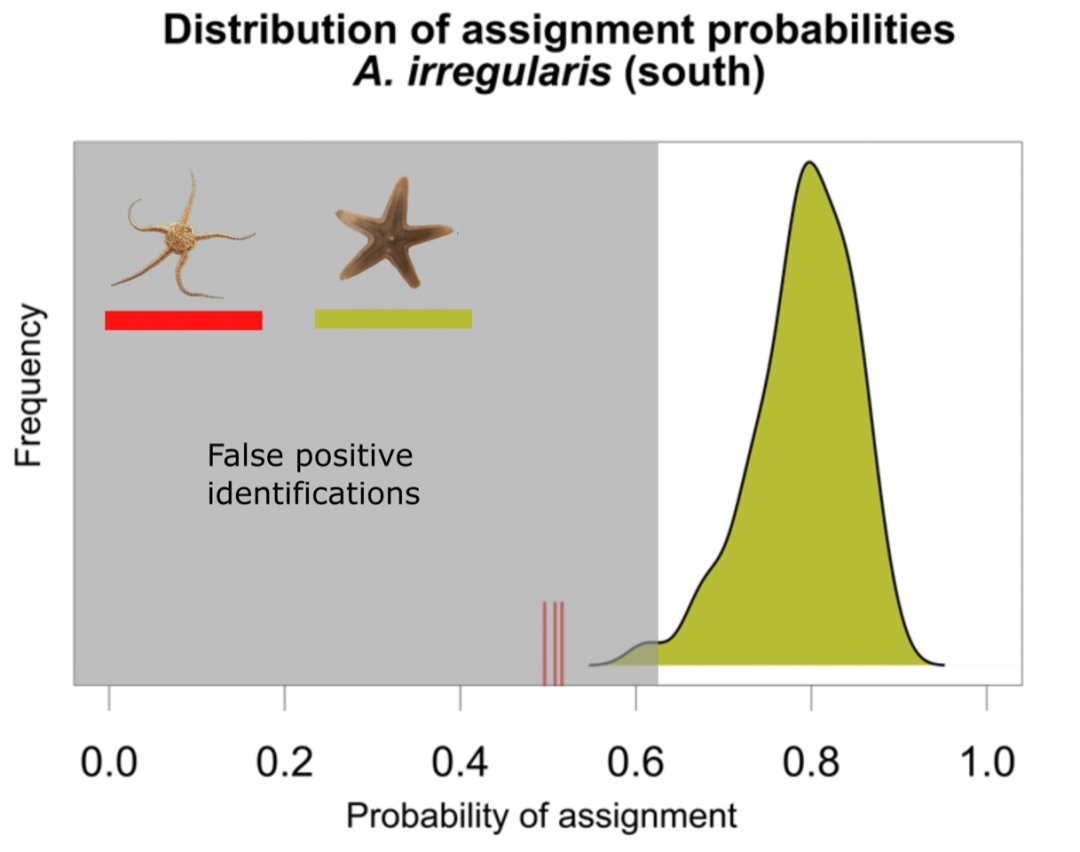
Wichtig für den zukünftigen Einsatz dieser Methode in ökologischen Arbeiten und der Biodiversitätserfassung ist das Vorhandensein entsprechender Analysetechniken. Dabei kommt es unter anderem darauf an, dass Organismen auf der Basis von Referenzdatenbanken identifiziert werden können. Wir arbeiten daran offen zugängliche Softwarelösungen für das Arbeiten mit Massenspektrometriedaten zu erstellen.
So konnten wir mittels Random Forest, einem Machine Learning Tool, bereits eine Lösung für Datenbankbasierte Identifikationen erarbeiten, die es ermöglicht nicht in der Referenzdatenbank vorhandene Arten zu erkennen, um so eine falsch positive Identifikation zu verhindern. Im Weiteren wird der Fokus darauf liegen, weitere Werkzeuge wie die automatische Ermittlung von Biodiversitätsindices zu erstellen um auch in Bereichen ohne Vergleichsdatenbanken ökologische Erfassungen zu ermöglichen.
Bereits durchgeführte Biodiversitätserfassungen
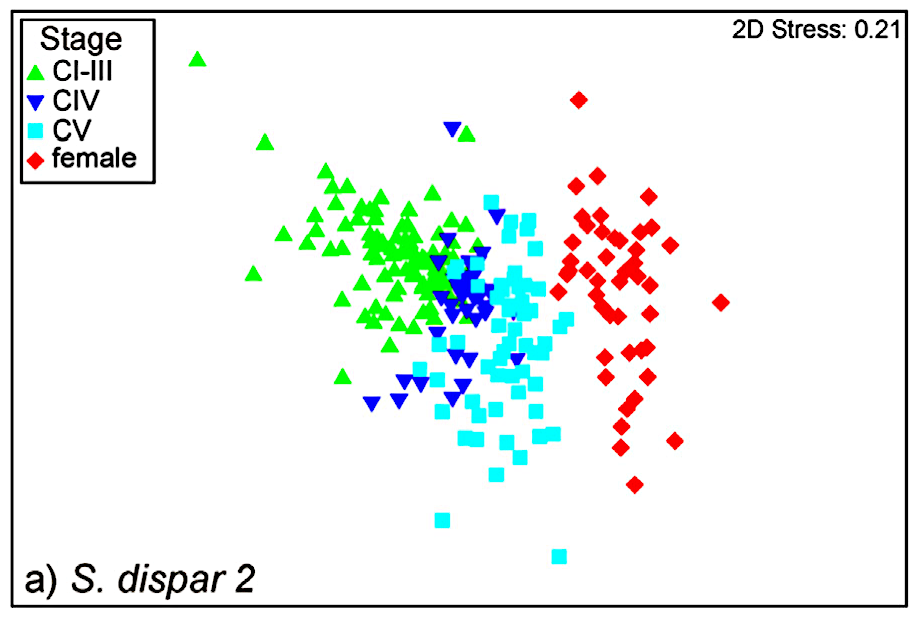
Neben der Methodenentwicklung und Pilotstudien haben wir bereits Biodiversitätserfassungen mittels MALDI-TOF MS durchgeführt. Hierbei wird der Protein-Fingerabdruck für eine große Anzahl Organismen gemessen um mit der darauf basierenden Artidentifikation Aussagen über die Lebensgemeinschaften dieser Tiere treffen zu können. In ersten Studien zeigte sich, dass die mit MALDI-TOF MS erfassten Lebensgemeinschaften hochgradig mit morphologisch bzw. molekulargenetisch erfassten Lebensgemeinschaften übereinstimmen. Dadurch konnte die Geschwindigkeit der Erfassung der untersuchten Tiere um ein vielfaches beschleunigt werden. So konnte z.B. bei spinocalaniden Copepoden aus der Tiefsee gezeigt werden, dass die vertikale Unterteilung von Habitaten eine große Rolle bei der reproduktiven Isolation und Koexistenz von kryptischen Arten spielt.
In zukünftigen Projekten soll die Methode zur Untersuchung der individuenreichen Meiofaunagemeinschaften der Nordsee genutzt werden. Auch die Meiofaunagesellschaften in der CCZ im Nordpazifik, einem Gebiet in dem es möglicher Tiefseebergbau an Manganknollen geben könnte, sollen mithilfe des Protein-Fingerabdrucks untersucht werden. Durch die Nutzung von MALDI-TOF MS werden wissenschaftliche Kapazitäten zur Erschließung neuer Arbeitsgebiete frei, die sonst durch die zeitaufwändige, morphologische Datenerfassung gebunden waren.
Abbildungsquellen
Abbildung Probenzustand.png: Rossel, S. & Martínez Arbizu, P. (2018). Effects of Sample Fixation on Specimen Identification in Biodiversity Assemblies based on Proteomic Data (MALDI-TOF). Frontiers in Marine Science, 5, 149.
Abbildung Pilotstudien.tif : Holst, S., Heins, A. & Laakmann, S. (2019). Morphological and molecular diagnostic species characters of Staurozoa (Cnidaria) collected on the coast of Helgoland (German Bight, North Sea). Marine Biodiversity. Retrieved from https://doi.org/10.1007/s12526-019-00943-1
Abbildungstext Abbildung Biodiversitätserfassungen.png: Kaiser, P., Bode, M., Cornils, A., Hagen, W., Arbizu, P.M., Auel, H. & Laakmann, S. (2018). High-resolution community analysis of deep-sea copepods using MALDI-TOF protein fingerprinting. Deep Sea Research Part I: Oceanographic Research Papers.